Koronavirus
Koronavirus atau virus korona ialah sekumpulan virus berkaitan yang menyebabkan penyakit dalam kalangan haiwan mamalia dan burung. Virus-virus ini berupaya menjangkiti manusia bermula dengan simptom-simptom jangkitan saluran pernafasan yang ringan seperti selesema biasa (majoriti disebabkan oleh rinovirus), sehingga penyakit yang boleh membunuh seperti SARS, MERS, dan COVID-19. Gejala-gejala yang dinampakkan oleh virus korona berbeza-beza mengikut spesies yang dijangkit contohnya penyakit salur pernafasan atas untuk ayam, dan cirit-birit untuk lembu dan khinzir.
| Orthocoronavirinae | |
|---|---|
| |
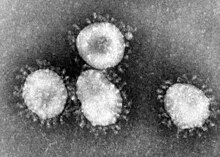
| Mikrograf elektron bagi virus bronkitis berjangkit virion | |

| |
| Ilustrasi morfologi koronavirus;
bentuk belantan pepaku peplomer virus, berwarna merah, menghasilkan rupa korona yang membungkus virion, apabila dilihat pada mikroskop elektron | |
| Pengelasan virus | |
| Templat taksonomi hilang (baiki): | '''Orthocoronavirinae''' |
| Genera[1] | |
| Sinonim[2][3][4] | |
| |
Koronavirus membentuk satu subfamili yang dikenali sebagai Orthocoronavirinae daripada famili Coronaviridae, order Nidovirales, dan alam Riboviria.[5][6] Ia diliputi virus dengan genom RNA bebenang tunggal positif dan nukleokapsid bersimetri heliks.[7] Ukuran genom koronavirus berkisar antara 26 hingga 32 kilobes, salah satu yang terbesar di antara virus RNA.[8] Virus ini mempunyai pancang berbentuk kelabu yang menonjol dari permukaannya, yang akan membentuk gambar korona suria di bawah mikrograf elektron, yang membentuk penamaannya sebagai koronavirus.[9]
Etimologi
suntingNama "koronavirus" (bahasa Inggeris: coronavirus) mengimbuhkan kata Latin corona bermaksud "mahkota"[10] oleh June Almeida dan David Tyrrell; ia dicipta menggambarkan ciri khas virion (bentuk infektif virus) koronavirus manusia yang pertama kali diperhatikan melalui mikroskop elektron di mana luar permukaannya mempunyai unjuran yang besar dan tumpul (bulir protein yang melekap pada permukaan virus[11]) seakan sinaran lingkar matahari.[9][12] Pemerhatian ini pertama kali tercatat pada tahun 1968 oleh sekumpulan ahli virologi tidak rasmi dalam jurnal Nature menamakan suatu famili baru virus yang berkongsi ciri luaran sama.[9]
Sejarah
suntingLaporan paling awal mengenai jangkitan koronavirus pada haiwan terjadi pada akhir 1920-an, ketika jangkitan pernafasan akut dari ayam peliharaan yang muncul di Amerika Utara.[13] Arthur Schalk dan M.C. Hawn pada tahun 1931 membuat laporan terperinci pertama yang menggambarkan jangkitan pernafasan ayam baru di Dakota Utara. Jangkitan anak ayam yang baru lahir disifatkan oleh tercungap-cungap dan kelesuan dengan kadar kematian yang tinggi 40-90%.[14] Leland David Bushnell dan Carl Alfred Brandly mengasingkan virus yang menyebabkan jangkitan pada tahun 1933.[15] Virus itu kemudian dikenali sebagai virus bronkitis berjangkit (IBV). Charles D. Hudson dan Fred Robert Beaudette mengulturkan virus itu untuk pertama kalinya pada tahun 1937.[16] Spesimen ini dikenali sebagai strain Beaudette. Pada akhir 1940-an, dua lagi koronavirus haiwan, JHM yang menyebabkan penyakit otak (murin ensefalitis) dan virus hepatitis tikus (MHV) yang menyebabkan hepatitis pada tikus ditemui.[17] Pada masa itu tidak disedari bahawa ketiga-tiga virus yang berlainan ini berkaitan.[18]
Koronavirus manusia ditemui pada tahun 1960[19][20] menggunakan dua kaedah yang berbeza di United Kingdom dan Amerika Syarikat.[21] E.C. Kendall, Malcolm Bynoe, dan David Tyrrell yang bekerja di Unit Selsema Biasa Majlis Penyelidikan Perubatan British mengumpulkan virus selesema biasa yang dinamakan B814 pada tahun 1961.[22][23][24] Virus ini tidak dapat dibiakkan menggunakan teknik piawai yang sama seperti teknik dalam membiakkan rinovirus, adenovirus dan virus selesema lain yang biasa. Pada tahun 1965, Tyrrell dan Bynoe berjaya membiakkan virus novel dengan menyebarkannya secara bersiri melalui kultur organ trakea embrio manusia.[25] Kaedah pembiakan baru diperkenalkan di makmal oleh Bertil Hoorn.[26] Virus yang telah diasingkan ketika diinokulasi secara intranasal secara tiba-tiba menyebabkan selesema dan dinyahaktifkan oleh eter yang menunjukkan bahawa ia mempunyai kurungan lipid.[22][27] Dorothy Hamre[28] dan John Procknow di University of Chicago mengasingkan novel selesema dari pelajar perubatan pada tahun 1962. Mereka mengasingkan dan mengembangkan virus dalam kultur tisu ginjal, menetapkannya sebagai 229E. Virus novel ini menyebabkan selesema secara tiba-tiba dan tidak diaktifkan oleh eter sama seperti B814.[29]
Ahli virologi Scotland, June Almeida di St Thomas Hospital, London, bekerjasama dengan Tyrrell, membandingkan struktur IBV, B814 dan 229E pada tahun 1967.[30][31] Dengan menggunakan mikroskopi elektron ketiga-tiga virus tersebut terbukti berkaitan secara morfologi dengan bentuk umum dan pancang berbentuk belantan.[32] Kumpulan penyelidikan di Institut Kesihatan Nasional pada tahun yang sama dapat mengasingkan ahli virus kumpulan baru ini menggunakan kultur organ dan menamakan salah satu sampel OC43 (OC untuk kultur organ).[33] Seperti B814, 229E, dan IBV, virus selesema novel OC43 mempunyai lonjakan seperti pancang berbentuk belantan yang khas ketika diperhatikan dengan mikroskop elektron.[34][35]
Virus selesema seperti IBV kemudiannya menunjukkan morfologinya yang berkaitan dengan virus hepatitis tikus.[17] Kumpulan virus baru ini diberi nama koronavirus disebabkan penampilan morfologi yang tersendiri.[9] Koronavirus manusia 229E dan koronavirus manusia OC43 terus dikaji dalam beberapa dekad berikutnya.[36][37] Strain koronavirus B814 hilang. Tidak diketahui penampilan seperti koronavirus manusia manakah strain tersebut.[38] Koronavirus manusia lain sejak itu dikenal pasti, termasuk SARS-CoV pada tahun 2003, HCoV NL63 pada tahun 2003, HCoV HKU1 pada tahun 2004, MERS-CoV pada tahun 2013, dan SARS-CoV-2 pada tahun 2020.[39] Terdapat juga sebilangan besar koronavirus haiwan yang dikenal pasti sejak tahun 1960-an.[5]
Mikrobiologi
suntingStruktur
suntingKoronavirus adalah zarah sfera pleomorfik besar dan kasar dengan unjuran permukaan yang unik.[40] Ukurannya sangat berubah-ubah dan diameter puratanya secara umum adalah 120 nm. Ukuran melampau diketahui dengan diameter 50 hingga 200 nm.200 nm.[41] Jisim keseluruhan molekul rata-rata 40,000 kDa. Ianya dibungkus oleh kurungan yang disertakan dengan sejumlah molekul protein.[42] Dwilapisan kurungan/sampul lipid, protein membran, dan nukleokapsid melindungi virus ketika berada di luar sel perumah.[43]
Sampul virus terdiri daripada dwilapisan lipid, yang menambat protein struktur membran (M), sampul (E) dan pancang (S).[44] Nisbah E:S:M dalam dwilapisan lipid adalah kira-kira 1:20:300.[45] Protein E dan M adalah protein struktur yang digabungkan dengan lapisan dwilipid membentuk sampul virus dan mengekalkan saiznya.[46] Protein S diperlukan untuk interaksi dengan sel perumah. Tetapi koronavirus manusia NL63 adalah pelik kerana protein M pada virus itu mempunyai tempat ikatan sel perumah, dan bukannya pada protein S.[47] Diameter sampul virus adalah 85 nm. Sampul virus dalam mikrograf elektron muncul sebagai sepasang cangkerang padat elektron (cangkang yang agak legap dengan sinar elektron yang digunakan untuk mengimbas zarah virus).[48][46]
Protein M adalah protein struktur utama sampul virus yang memberikan bentuk keseluruhan dan merupakan protein membran jenis ketiga (III). Ia terdiri daripada 218 hingga 263 sisa asid amino dan membentuk lapisan dengan ketebalan 7.8 nm.[42] Ia mempunyai tiga domain seperti ektodomain terminus-N pendek, domain transmembran tiga rentang, dan endodomain terminus-C. Domain terminus-C membentuk kekisi seperti matriks yang menambah ketebalan sampul virus tambahan. Spesies yang berlainan boleh mempunyai glikans bersambung dengan -N atau -O dalam domain terminal amino protein mereka. Protein M sangat penting dalam kitaran hidup virus seperti semasa pemasangan, penunasan, pembentukan sampul, dan patogenesis.[49]
Protein E adalah protein struktur kecil dan sangat selalu berubah-ubah dalam spesies yang berbeza. Hanya terdapat sekitar 20 protein E dalam koronavirus. Ia berukuran 8.4 hingga 12 kDa dan terdiri daripada 76 hingga 109 asid amino. [41] Ia adalah protein integral (iaitu tertanam di lapisan lipid) dan mempunyai dua domain iaitu domain transmembran dan domain terminal-ekstramembrannya hampir sepenuhnya α-heliks, dengan domain transmembran α-helikal tunggal, dan membentuk liang ion pentamerik (lima molekul) di lapisan dwilipid. Mereka bertanggungjawab untuk pemasangan virion, pengangkutan intraselular dan morfogenesis (penunasan).[42]
Pancang adalah ciri koronavirus yang paling berbeza, dan bertanggungjawab untuk pembentukan permukaan seperti korona atau halo. Rata-rata zarah koronavirus mempunyai 74 pancang pada permukaan.[50] Setiap pancang panjangnya kira-kira 20 nm dan terdiri daripada trimer protein S. Protein S seterusnya terdiri daripada subunit S1 dan S2. Protein homotrimerik S adalah pelakuran protein kelas satu (I) yang menjadi pengantara pengikatan reseptor dan pelakuran membran antara virus dan sel perumah. Subunit S1 membentuk kepala pancang dan mempunyai domain pengikat reseptor (RBD). Subunit S2 membentuk batang yang menambat pancang sampul virus dan pengaktifan protease memungkinkan penyatuan. Kedua-dua subunit tersebut tetap dihubungkan secara tidak kovalen kerana mereka terdedah pada permukaan virus, sehingga melekat pada membran sel perumah.[42] Dalam keadaan aktif berfungsi, tiga S1 dilampirkan pada dua subunit S2. Kompleks subunit dibahagikan kepada subunit individu apabila virus mengikat dan menyatu dengan sel perumah di bawah tindakan protease seperti famili katepsin dan transmembran protease serina 2 (TMPRSS2) sel perumah.[51]
Protein S1 adalah komponen yang paling kritikal dari segi jangkitan. Mereka juga merupakan komponen yang paling berubah-ubah kerana mereka bertanggungjawab untuk pengkhususan sel perumah. Mereka memiliki dua domain utama bernama domain N-terminal (S1-NTD) dan domain C-terminal (S1-CTD), yang keduanya berfungsi sebagai domain pengikat reseptor. NTD mengenali dan mengikat gula di permukaan sel perumah. Pengecualian adalah MHV NTD yang mengikat pada protein molekul lekatan sel berkait dengan antigen karsinoembrionik 1 reseptor protein (CEACAM1). S1-CTDs bertanggungjawab untuk mengenalpasti reseptor protein yang berbeza seperti enzim penukaran angiotensin 2 (ACE2), aminopeptidase N (APN), dan dipeptidil peptidase 4 (DPP4).[42]
Sebilangan kecil koronavirus (khususnya anggota subkumpulan A betacoronavirus) juga mempunyai protein permukaan seperti pancang yang lebih pendek yang disebut hemaglutinin esterase (HE).[5] Protein HE bertindak sebagai homodimer yang terdiri daripada sekitar 400 sisa asid amino dan berukuran 40 hingga 50 kDa. Mereka muncul sebagai unjuran permukaan kecil sepanjang 5 hingga 7 nm yang tertanam di antara pancang. Mereka membantu melekat dan melepaskan diri dari sel perumah.[52]
Di dalam sampul virus itu, terdapat [[kapsid|nukleokapsid], yang terbentuk dari beberapa salinan protein nukleokapsid (N), yang terikat pada genom RNA bebenang tunggal positif dalam bentuk jenis seperti manik-pada-tali secara berterusan.[46][53] Protein N adalah fosfoprotein berukuran 43 hingga 50 kDa, dan dibahagikan kepada tiga domain terpelihara. Sebilangan besar protein terdiri daripada domain 1 dan 2, yang biasanya kaya dengan arginina dan lisina. Domain 3 mempunyai hujung terminal karboksi pendek dan mempunyai cas negatif bersih disebabkan oleh lebihan asid berbanding sisa asid amino bes.[41]
Genom
suntingKesemua virus korona mempunyai genom RNA bererti-positif dan bebenang tunggal. Saiz genom koronavirus lazimnya di antara 27 dan 34 kilobes.[8] Saiz genom virus ini merupakan antara yang terbesar di kalangan virus-virus RNA. Genom ini mempunyai satu kepala 5-prima (5') bermetil dan satu ekor 3-prima (3') berpoliadenil.[54]
Genom koronavirus ini mempunyai organisasi yang berstruktur kepala 5' dengan kawasan tak ditranslasi (kepala 5′-UTR)-replikase/transkriptase-bulir/pepaku (S)-sampul (E)-membran (M)-nukleokapsid (N)- ekor 3' UTR poli-A. Dua per tiga pertama daripada genom virus ini adalah terdiri daripada rangka bacaan terbuka 1a dan 1b, yang mengekod poliprotein replikase dan transkriptase. Poliprotein replikase dan transkriptase terbelah secara sendiri untuk membentuk protein tidak berstruktur (nsps).[55]
Rangka bacaan di bahagian belakang pula mengekod empat protein berstruktur terbesar yakni protein pepaku, protein sampul, protein membran, dan protein nukleokapsid.[56] Rangka-rangka bacaan lain yang terapit di antara rangka-rangka tersebut adalah rangka bacaan untuk protein-protein hiasan lain. Bilangan protein-protein hiasan dan fungsinya adalah unik bergantung kepada jenis-jenis koronavirus.[55]
Kitaran hidup
suntingKemasukan
suntingJangkitan bermula apabila glikoprotein pepaku (S) virus ini bersambung dengan pelengkap reseptor sel perumah. Selepas penyambungan berjaya, protease dari sel perumah akan terbelah dan mengaktifkan protein pepaku yang tersambung. Pembelahan dan pengaktifan ini membenarkan kemasukan virus ke dalam sel perumah melalui sama ada endositosis atau penyatuan langsung antara envelop virus dengan membran perumah, namun proses ini bergantung kepada ketersediaan protease sel perumah. [57]
Sewaktu virus ini memasuki sel perumah, zarah virus ini dinyahlapis dan kandungan genom virus ini memasuki sitoplasma sel.[58] Genom RNA koronavirus mempunyai kepala 5' bermetil dan ekor 3' berpoliadenil yang membenarkan RNA virus bergabung dengan ribosom sel perumah untuk proses translasi.[59] Ribosom dari perumah mentranslasi bahagian permulaan dari rangka bacaan terbuka genom virus yang bertindan dan membentuk poliprotein yang panjang. Poliprotein ini juga mempunyai protease tersendiri yang berupaya untuk membelah poliprotein kepada pelbagai protein tak berstruktur.[55]
Pereplikaan
suntingSebahagian dari protein-protein tak berstruktur ini akan bertaut untuk membentuk kompleks multiprotein replikase-transkriptase (RTC). Protein replikase-transkriptase yang utama ialah polimerase RNA yang bergantung pada RNA (RdRp). Ianya terlibat secara langsung dalam pereplikaan dan pentranskriptaan RNA dari seutas RNA. Protein-protein tidak berstruktur daripada kompleks tersebut turut terlibat dalam proses replikasi dan transkriptasi. Contohnya, eksoribonuklease (yang juga merupakan sebahagian daripada protein tak berstruktur) terlibat dalam penyemakbetulan rantaian RNA yang direplikasi. Proses ini adalah penting kerana polimerase RNA bergantung pada RNA tidak dapat melakukan proses ini.[60]
Salah satu dari fungsi utama kompleks ini adalah mereplikasi genom virus ini. RdRp secara langsung mengantara sintesis genom RNA bererti-negatif dari genom RNA bererti-positif, dan kemudiannya mensintesis semula genom RNA bererti-positif dari templat genom RNA bererti-negatif.[55] Satu lagi fungsi lain yang penting bagi kompleks ini adalah mentranskript genom virus. RdRp mengantara secara langsung sintesis molekul-molekul subgenom RNA bererti-negatif dari genom RNA bererti-positif, yang dikuti dengan transkripsi molekul-molekul RNA ini kepada pengutus RNA (mRNA) bererti-positif.[55]
Pembebasan
suntingGenom RNA bererti-positif yang tereplikasi menjadi genom virus-virus progeni. mRNA-mRNA ini merupakan transkrip-transkrip gen daripada sepertiga terakhir genom virus selepas rangka bacaan awal yang bertindan. Mereka ditranslasi oleh ribosom-ribosom perumah kepada protein-protein berstruktur dan sebilangan dari protein-protein aksesori.[55] Translasi RNA berlaku dalam retikulum endoplasma. Protein-protein viral berstruktur S, E dan M bergerak melalui laluan perembesan ke dalam kompartmen sederhana Golgi. Di situ juga, protein-protein M mengawal kebanyakan interaksi-interaksi antara protein yang diperlukan untuk pembentukan virus-virus baharu selepas protein-protein M ini berpaut kepada nukleokapsid.[61] Virus-virus progeni ini dibebaskan melalui vesikel rembesan dari sel-sel perumah melalui proses eksositosis.[61]
Virus-virus korona manusia
suntingVirus-virus korona berbeza antara satu sama lain dengan ketara dari segi faktor risiko. Sesetengah daripada virus korona boleh menyebabkan penyakit yang membunuh lebih daripada 30% pesakit yang dijangkiti (contohnya MERS-CoV), dan penyakit bagi sesetengah virus lain pula secara relatifnya adalah tidak berbahaya, seperti selesema biasa.[46] Virus-virus ini berupaya untuk menyebabkan selesema dengan gejala-gejala utama seperti demam dan sakit tekak yang berpunca daripada adenoid yang bengkak, dan kebiasaannya jangkitan berlaku pada musim sejuk dan awal musim bunga.[62] Koronavirus boleh menyebabkan pneumonia (sama ada secara langsung daripada virus ataupun jangkitan sekunder dari bakteria) dan bronkitis (juga boleh diperolehi secara langsung daripada virus mahupun secara sekunder dari bakteria).[63] SARS-CoV, sejenis koronavirus manusia yang banyak dikemukakan dalam dunia perubatan ditemui pada tahun 2003, yang juga penyebab sindrom pernafasan akut teruk (SARS), mempunyai ciri patogenesis yang unik kerana ia menyebabkan jangkitan-jangkitan trakus pernafasan atas dan bawah.[63]
Terdapat enam jenis virus-virus korona yang diketahui dengan salah satu daripada spesies-spesies tersebut terbahagi kepada dua sub-strain yang berbeza, menjadikan jumlah strain secara keseluruhannya tujuh. Empat daripada stain-strain ini menyebabkan gejala-gejala selesema biasa yang ringan:
- Koronavirus manusia OC43 (HCoV-OC43), daripada genus β-CoV
- Koronavirus manusia HKU1, β-CoV, mempunyai persamaan genom dengan OC43 sebanyak 75%[64]
- Koronavirus manusia 229E (HCoV-229E), α-CoV
- Koronavirus manusia NL63 (HCoV-NL63, Koronavirus New Haven), α-CoV
– dan tiga lagi strain (daripada dua spesies) berupaya untuk menyebabkan gejala-gejala yang teruk, dan ketiga-tiga strain ini adalah daripada kumpulan strain β-CoV:
- Koronavirus berkaitan sindrom pernafasan Timur Tengah (MERS-CoV), sebelum ini dikenali dengan nama koronavirus baharu 2012 (novel coronavirus 2012) dan HCoV-EMC
- Koronavirus berkaitan sindrom pernafasan akut teruk (SARS-CoV or "klasik SARS")
- Koronavirus sindrom pernafasan akut teruk 2 (SARS-CoV-2), sebelum ini dikenali dengan nama 2019-nCoV atau "koronavirus baharu 2019"
Virus-virus korona HCoV-229E, -NL63, -OC43, and -HKU1 menyebar secara berterusan di kalangan populasi manusia dan menyebabkan jangkitan-jangkitan pernafasan secara berterusan di kalangan orang dewasa dan kanak-kanak serata dunia.[65]
Lihat juga
sunting- Zoonosis
- Virus bawaan kelawar
- Koronavirus manusia HKU1
- Koronavirus manusia OC43
- Koronavirus manusia 229E
- MERS-CoV
- Koronavirus kelawar Rhinolophus HKU2
- SADS-CoV
- SARS-CoV
- Koronavirus kelawar Pipistrellus HKU5
- Koronavirus kelawar Tylonycteris HKU4
- Koronavirus kelawar Rousettus HKU9
- Bat SL-CoV-WIV1
- London1 novel CoV/2012
- HCoV-EMC/2012
- 2019-nCoV
Rujukan
sunting- ^ "Virus Taxonomy: 2018b Release". International Committee on Taxonomy of Viruses (ICTV) (dalam bahasa Inggeris). March 2019. Diarkibkan daripada yang asal pada 4 March 2018. Dicapai pada 24 January 2020.
- ^ "2017.012-015S" (xlsx). International Committee on Taxonomy of Viruses (ICTV) (dalam bahasa Inggeris). October 2018. Diarkibkan daripada yang asal pada 14 May 2019. Dicapai pada 24 January 2020.
- ^ Fan Y, Zhao K, Shi ZL, Zhou P (March 2019). "Bat Coronaviruses in China". Viruses. 11 (3): 210. doi:10.3390/v11030210. PMC 6466186. PMID 30832341.
- ^ a b c de Groot RJ, Baker SC, Baric R, Enjuanes L, Gorbalenya AE, Holmes KV, Perlman S, Poon L, Rottier PJ, Talbot PJ, Woo PC, Ziebuhr J (2011). "Family Coronaviridae". Dalam King AM, Lefkowitz E, Adams MJ, Carstens EB, International Committee on Taxonomy of Viruses, International Union of Microbiological Societies. Virology Division (penyunting). Ninth Report of the International Committee on Taxonomy of Viruses. Oxford: Elsevier. m/s. 806–28. ISBN 978-0-12-384684-6. Ralat petik: Tag
<ref>tidak sah, nama "groot" digunakan secara berulang dengan kandungan yang berbeza - ^ International Committee on Taxonomy of Viruses (24 August 2010). "ICTV Master Species List 2009—v10" (xls).
- ^ Cherry, James; Demmler-Harrison, Gail J.; Kaplan, Sheldon L.; Steinbach, William J.; Hotez, Peter J. (2017). Feigin and Cherry's Textbook of Pediatric Infectious Diseases (dalam bahasa Inggeris). Elsevier Health Sciences. m/s. PT6615. ISBN 978-0-323-39281-5.
- ^ a b Woo PC, Huang Y, Lau SK, Yuen KY (August 2010). "Coronavirus genomics and bioinformatics analysis". Viruses. 2 (8): 1804–20. doi:10.3390/v2081803. PMC 3185738. PMID 21994708.
Coronaviruses possess the largest genomes [26.4 kb (ThCoV HKU12) to 31.7 kb (SW1)] among all known RNA viruses (Figure 1) [2,13,16].
- ^ a b c d Almeida JD, Berry DM, Cunningham CH, Hamre D, Hofstad MS, Mallucci L, McIntosh K, Tyrrell DA (November 1968). "Virology: Coronaviruses". Nature. 220 (5168): 650. Bibcode:1968Natur.220..650.. doi:10.1038/220650b0.
[T]here is also a characteristic "fringe" of projections 200 A long, which are rounded or petal shaped ... This appearance, recalling the solar corona, is shared by mouse hepatitis virus and several viruses recently recovered from man, namely strain B814, 229E and several others.
- ^ "cŏrōna". K. Prent, J. Adisubrata, W. J. S. Poerwadarminta (1969). Kamus Latin-Indonesia. Jogjakarta: Penerbitan Jajasan Kanisius. m/s. 198.
- ^ Sturman LS, Holmes KV (Jan 1983). Lauffer MA, Maramorosch K (penyunting). "The molecular biology of coronaviruses". Advances in Virus Research. 28: 35–112. doi:10.1016/s0065-3527(08)60721-6. ISBN 9780120398287. PMC 7131312. PMID 6362367.
[T]hese viruses displayed a characteristic fringe of large, distinctive, petal-shaped peplomers or spikes which resembled a crown, like the corona spinarum in religious art; hence the name coronaviruses.
- ^ Tyrrell, David Arthur John; Fielder, Michael (2002). Cold Wars: The Fight Against the Common Cold (dalam bahasa Inggeris). Oxford University Press. m/s. 96. ISBN 978-0-19-263285-2.
We looked more closely at the appearance of the new viruses and noticed that they had a kind of halo surrounding them. Recourse to a dictionary produced the Latin equivalent, corona, and so the name coronavirus was born.
Unknown parameter|name-list-format=ignored (bantuan) - ^ Estola T (1970). "Coronaviruses, a New Group of Animal RNA Viruses". Avian Diseases. 14 (2): 330–336. doi:10.2307/1588476. ISSN 0005-2086. JSTOR 1588476. PMID 4316767.
- ^ Fabricant, Julius (1998). "The Early History of Infectious Bronchitis". Avian Diseases. 42 (4): 648–650. doi:10.2307/1592697. ISSN 0005-2086. JSTOR 1592697. PMID 9876830. Unknown parameter
|name-list-format=ignored (bantuan) - ^ Bushnell LD, Brandly CA (1933). "Laryngotracheitis in chicks". Poultry Science (dalam bahasa Inggeris). 12 (1): 55–60. doi:10.3382/ps.0120055.
- ^ Decaro, Nicola (2011). "Gammacoronavirus". Dalam Tidona, Christian; Darai, Gholamreza (penyunting). Gammacoronavirus‡: Coronaviridae. The Springer Index of Viruses (dalam bahasa Inggeris). Springer. m/s. 403–413. doi:10.1007/978-0-387-95919-1_58. ISBN 978-0-387-95919-1. PMC 7176155. Unknown parameter
|name-list-format=ignored (bantuan) - ^ a b McIntosh K (1974). "Coronaviruses: A Comparative Review". Dalam Arber W, Haas R, Henle W, Hofschneider PH, Jerne NK, Koldovský P, Koprowski H, Maaløe O, Rott R (penyunting). Current Topics in Microbiology and Immunology / Ergebnisse der Mikrobiologie und Immunitätsforschung. Current Topics in Microbiology and Immunology / Ergebnisse der Mikrobiologie und Immunitätsforschung (dalam bahasa Inggeris). Berlin, Heidelberg: Springer. m/s. 87. doi:10.1007/978-3-642-65775-7_3. ISBN 978-3-642-65775-7.
- ^ "Il était une fois les coronavirus". Réalités Biomédicales (dalam bahasa Perancis). 2020-03-27. Dicapai pada 2020-04-18.
- ^ Kahn JS, McIntosh K (November 2005). "History and recent advances in coronavirus discovery". The Pediatric Infectious Disease Journal. 24 (11 Suppl): S223–7, discussion S226. doi:10.1097/01.inf.0000188166.17324.60. PMID 16378050.
- ^ Mahase E (April 2020). "The BMJ in 1965". BMJ. 369: m1547. doi:10.1136/bmj.m1547. PMID 32299810.
- ^ Monto, Arnold S. (1984). "Coronaviruses". Dalam Evans, Alfred S. (penyunting). Viral Infections of Humans. Viral Infections of Humans: Epidemiology and Control (dalam bahasa Inggeris). Springer US. m/s. 151–165. doi:10.1007/978-1-4684-4727-9_7. ISBN 978-1-4684-4727-9. Unknown parameter
|name-list-format=ignored (bantuan) - ^ a b Kendall EJ, Bynoe ML, Tyrrell DA (July 1962). "Virus isolations from common colds occurring in a residential school". British Medical Journal. 2 (5297): 82–6. doi:10.1136/bmj.2.5297.82. PMC 1925312. PMID 14455113.
- ^ Richmond, Caroline (2005-06-18). "David Tyrrell". BMJ : British Medical Journal. 330 (7505): 1451. doi:10.1136/bmj.330.7505.1451. PMC 558394. Unknown parameter
|name-list-format=ignored (bantuan) - ^ "Obituary Notices: Malcom Byone". British Medical Journal (dalam bahasa Inggeris). 2 (5660): 827–829. 1969-06-28. doi:10.1136/bmj.2.5660.827. S2CID 220187042.
- ^ Tyrrell DA, Bynoe ML (June 1965). "Cultivation of a Novel Type of Common-Cold Virus in Organ Cultures". British Medical Journal. 1 (5448): 1467–70. doi:10.1136/bmj.1.5448.1467. PMC 2166670. PMID 14288084.
- ^ Tyrrell, David Arthur John; Fielder, Michael (2002). Cold Wars: The Fight Against the Common Cold (dalam bahasa Inggeris). Oxford University Press. m/s. 93–95. ISBN 978-0-19-263285-2. Unknown parameter
|name-list-format=ignored (bantuan) - ^ Hagan, William Arthur; Bruner, Dorsey William; Gillespie, James Howard; Timoney, John Francis; Scott, Fredric W.; Barlough, Jeffrey E. (1988). Hagan and Bruner's Microbiology and Infectious Diseases of Domestic Animals: With Reference to Etiology, Epizootiology, Pathogenesis, Immunity, Diagnosis, and Antimicrobial Susceptibility (dalam bahasa Inggeris). Cornell University Press. m/s. 440. ISBN 978-0-8014-1896-9. Unknown parameter
|name-list-format=ignored (bantuan) - ^ Knapp, Alex. "The Secret History Of The First Coronavirus". Forbes (dalam bahasa Inggeris). Dicapai pada 2020-05-06.
- ^ Hamre D, Procknow JJ (January 1966). "A new virus isolated from the human respiratory tract". Proceedings of the Society for Experimental Biology and Medicine. 121 (1): 190–3. doi:10.3181/00379727-121-30734. PMID 4285768. S2CID 1314901.
- ^ "The woman who discovered the first coronavirus".
- ^ Almeida, Joyce (2008-06-26). "June Almeida (née Hart)". BMJ (dalam bahasa Inggeris). 336 (7659): 1511.1–1511. doi:10.1136/bmj.a434. ISSN 0959-8138. PMC 2440895. Unknown parameter
|name-list-format=ignored (bantuan) - ^ Almeida JD, Tyrrell DA (April 1967). "The morphology of three previously uncharacterized human respiratory viruses that grow in organ culture". The Journal of General Virology. 1 (2): 175–8. doi:10.1099/0022-1317-1-2-175. PMID 4293939.
- ^ McIntosh K, Becker WB, Chanock RM (December 1967). "Growth in suckling-mouse brain of "IBV-like" viruses from patients with upper respiratory tract disease". Proceedings of the National Academy of Sciences of the United States of America. 58 (6): 2268–73. Bibcode:1967PNAS...58.2268M. doi:10.1073/pnas.58.6.2268. PMC 223830. PMID 4298953.
- ^ McIntosh K, Dees JH, Becker WB, Kapikian AZ, Chanock RM (April 1967). "Recovery in tracheal organ cultures of novel viruses from patients with respiratory disease". Proceedings of the National Academy of Sciences of the United States of America. 57 (4): 933–40. Bibcode:1967PNAS...57..933M. doi:10.1073/pnas.57.4.933. PMC 224637. PMID 5231356.
- ^ Times, Harold M. Schmeck Jr Special To the New York (1967-05-05). "Six Newly Discovered Viruses May Explain Cold; Strains Are Similar to Germ That Causes a Bronchial Infection in Chickens Believed to Be New Group". The New York Times (dalam bahasa Inggeris). ISSN 0362-4331. Dicapai pada 2020-04-25.
- ^ Myint, Steven H. (1995). "Human Coronavirus Infections". Dalam Siddell, Stuart G. (penyunting). The Coronaviridae. The Viruses (dalam bahasa Inggeris). Springer US. m/s. 389–401. doi:10.1007/978-1-4899-1531-3_18. ISBN 978-1-4899-1531-3. Unknown parameter
|name-list-format=ignored (bantuan) - ^ Geller C, Varbanov M, Duval RE (November 2012). "Human coronaviruses: insights into environmental resistance and its influence on the development of new antiseptic strategies". Viruses. 4 (11): 3044–68. doi:10.3390/v4113044. PMC 3509683. PMID 23202515.
- ^ Corman VM, Jores J, Meyer B, Younan M, Liljander A, Said MY, dll. (August 2014). "Antibodies against MERS coronavirus in dromedary camels, Kenya, 1992-2013". Emerging Infectious Diseases. 20 (8): 1319–22. doi:10.1007/978-1-4899-7448-8_10. ISBN 978-1-4899-7447-1. PMC 7122465. PMID 25075637.
The other OC strains and B814 that could not be adapted to mouse brain resisted adaptation to cell culture as well; these distinct viruses have since been lost and may actually have been rediscovered recently.
- ^ Zhu N, Zhang D, Wang W, Li X, Yang B, Song J, dll. (February 2020). "A Novel Coronavirus from Patients with Pneumonia in China, 2019". The New England Journal of Medicine. 382 (8): 727–733. doi:10.1056/NEJMoa2001017. PMC 7092803. PMID 31978945.
- ^ Goldsmith CS, Tatti KM, Ksiazek TG, Rollin PE, Comer JA, Lee WW, dll. (February 2004). "Ultrastructural characterization of SARS coronavirus". Emerging Infectious Diseases. 10 (2): 320–26. doi:10.3201/eid1002.030913. PMC 3322934. PMID 15030705.
Virions acquired an envelope by budding into the cisternae and formed mostly spherical, sometimes pleomorphic, particles that averaged 78 nm in diameter (Figure 1A).
- ^ a b c Masters, Paul S. (2006). "The molecular biology of coronaviruses". Advances in Virus Research. 66: 193–292. doi:10.1016/S0065-3527(06)66005-3. ISBN 9780120398690. PMC 7112330. PMID 16877062.
- ^ a b c d e Lalchhandama, K. (2020). "The chronicles of coronaviruses: the electron microscope, the doughnut, and the spike". Science Vision (dalam bahasa Inggeris). 20 (2): 78–92. doi:10.33493/scivis.20.02.03.
- ^ Neuman BW, Kiss G, Kunding AH, Bhella D, Baksh MF, Connelly S, dll. (April 2011). "A structural analysis of M protein in coronavirus assembly and morphology". Journal of Structural Biology. 174 (1): 11–22. doi:10.1016/j.jsb.2010.11.021. PMC 4486061. PMID 21130884.
See Figure 10.
- ^ Lai MM, Cavanagh D (1997). "The molecular biology of coronaviruses". Advances in Virus Research. 48: 1–100. doi:10.1016/S0065-3527(08)60286-9. ISBN 9780120398485. PMC 7130985. PMID 9233431.
- ^ Cavanagh D, Mawditt K, Sharma M, Drury SE, Ainsworth HL, Britton P, Gough RE (August 2001). Schmidt A, Weber O, Wolff MH (penyunting). "Detection of a coronavirus from turkey poults in Europe genetically related to infectious bronchitis virus of chickens". Avian Pathology. Birkhäuser Advances in Infectious Diseases BAID. Birkhäuser. 30 (4): 355–68. doi:10.1007/3-7643-7339-3_1. ISBN 978-3-7643-7339-9. PMC 7123520. PMID 19184921.
- ^ a b c d Fehr AR, Perlman S (2015). "Coronaviruses: an overview of their replication and pathogenesis". Dalam Maier HJ, Bickerton E, Britton P (penyunting). Coronaviruses. Methods in Molecular Biology. 1282. Springer. m/s. 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466.
See section: Virion Structure.
- ^ Naskalska, Antonina; Dabrowska, Agnieszka; Szczepanski, Artur; Milewska, Aleksandra; Jasik, Krzysztof Piotr; Pyrc, Krzysztof (2019). "Membrane Protein of Human Coronavirus NL63 Is Responsible for Interaction with the Adhesion Receptor". Journal of Virology. 93 (19). doi:10.1128/JVI.00355-19. PMC 6744225. PMID 31315999.
- ^ Neuman BW, Adair BD, Yoshioka C, Quispe JD, Orca G, Kuhn P, dll. (August 2006). "Supramolecular architecture of severe acute respiratory syndrome coronavirus revealed by electron cryomicroscopy". Journal of Virology. 80 (16): 7918–28. doi:10.1128/JVI.00645-06. PMC 1563832. PMID 16873249.
Particle diameters ranged from 50 to 150 nm, excluding the spikes, with mean particle diameters of 82 to 94 nm; Also See Figure 1 for double shell.
- ^ Schoeman, Dewald; Fielding, Burtram C. (2019). "Coronavirus envelope protein: current knowledge". Virology Journal (dalam bahasa Inggeris). 16 (1): 69. doi:10.1186/s12985-019-1182-0. PMC 6537279. PMID 31133031.
- ^ Neuman, Benjamin W.; Kiss, Gabriella; Kunding, Andreas H.; Bhella, David; Baksh, M. Fazil; Connelly, Stephen; Droese, Ben; Klaus, Joseph P.; Makino, Shinji; Sawicki, Stanley G.; Siddell, Stuart G. (April 2011). "A structural analysis of M protein in coronavirus assembly and morphology". Journal of Structural Biology. 174 (1): 11–22. doi:10.1016/j.jsb.2010.11.021. ISSN 1047-8477. PMC 4486061. PMID 21130884.
- ^ Alsaadi, Entedar A. J.; Jones, Ian M. (2019). "Membrane binding proteins of coronaviruses". Future Virology. 14 (4): 275–286. doi:10.2217/fvl-2018-0144. PMC 7079996. PMID 32201500.
- ^ Zeng, Qinghong; Langereis, Martijn A.; van Vliet, Arno L. W.; Huizinga, Eric G.; de Groot, Raoul J. (2008). "Structure of coronavirus hemagglutinin-esterase offers insight into corona and influenza virus evolution". Proceedings of the National Academy of Sciences of the United States of America. 105 (26): 9065–9069. doi:10.1073/pnas.0800502105. PMC 2449365. PMID 18550812.
- ^ Chang CK, Hou MH, Chang CF, Hsiao CD, Huang TH (March 2014). "The SARS coronavirus nucleocapsid protein—forms and functions". Antiviral Research. 103: 39–50. doi:10.1016/j.antiviral.2013.12.009. PMC 7113676. PMID 24418573.
See Figure 4c.
- ^ Fehr AR, Perlman S (2015). Maier HJ, Bickerton E, Britton P (penyunting). "An Overview of Their Replication and Pathogenesis; Section 2 Genomic Organization". Methods in Molecular Biology. Springer. 1282: 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466. Unknown parameter
|name-list-format=ignored (bantuan) - ^ a b c d e f Fehr AR, Perlman S (2015). "Coronaviruses: an overview of their replication and pathogenesis". Methods in Molecular Biology. 1282: 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2437-0. PMC 4369385. PMID 25720466.
- ^ Snijder EJ, Bredenbeek PJ, Dobbe JC, Thiel V, Ziebuhr J, Poon LL, dll. (August 2003). "Unique and conserved features of genome and proteome of SARS-coronavirus, an early split-off from the coronavirus group 2 lineage". Journal of Molecular Biology. 331 (5): 991–1004. doi:10.1016/S0022-2836(03)00865-9. PMID 12927536.
See Figure 1.
- ^ Simmons G, Zmora P, Gierer S, Heurich A, Pöhlmann S (December 2013). "Proteolytic activation of the SARS-coronavirus spike protein: cutting enzymes at the cutting edge of antiviral research". Antiviral Research. 100 (3): 605–14. doi:10.1016/j.antiviral.2013.09.028. PMC 3889862. PMID 24121034.
See Figure 2.
- ^ Fehr, Anthony R.; Perlman, Stanley (2015), Maier, Helena Jane; Bickerton, Erica; Britton, Paul (penyunting), "Coronaviruses: An Overview of Their Replication and Pathogenesis; Section 4.1 Attachment and Entry", Coronaviruses: Methods and Protocols, Methods in Molecular Biology (dalam bahasa Inggeris), Springer, 1282, m/s. 1–23, doi:10.1007/978-1-4939-2438-7_1, ISBN 978-1-4939-2438-7, PMC 4369385, PMID 25720466 Unknown parameter
|name-list-format=ignored (bantuan) - ^ Fehr, Anthony R.; Perlman, Stanley (2015), Maier, Helena Jane; Bickerton, Erica; Britton, Paul (penyunting), "Coronaviruses: An Overview of Their Replication and Pathogenesis; Section 2 Genomic Organization", Coronaviruses: Methods and Protocols, Methods in Molecular Biology (dalam bahasa Inggeris), Springer, m/s. 1–23, doi:10.1007/978-1-4939-2438-7_1, ISBN 978-1-4939-2438-7, PMC 4369385, PMID 25720466 Unknown parameter
|name-list-format=ignored (bantuan) - ^ Sexton NR, Smith EC, Blanc H, Vignuzzi M, Peersen OB, Denison MR (August 2016). "Homology-Based Identification of a Mutation in the Coronavirus RNA-Dependent RNA Polymerase That Confers Resistance to Multiple Mutagens". Journal of Virology. 90 (16): 7415–28. doi:10.1128/JVI.00080-16. PMC 4984655. PMID 27279608.
Finally, these results, combined with those from previous work (33, 44), suggest that CoVs encode at least three proteins involved in fidelity (nsp12-RdRp, nsp14-ExoN, and nsp10), supporting the assembly of a multiprotein replicase-fidelity complex, as described previously (38).
- ^ a b Fehr AR, Perlman S (2015). "Coronaviruses: an overview of their replication and pathogenesis". Dalam Maier HJ, Bickerton E, Britton P (penyunting). Coronaviruses. Methods in Molecular Biology. 1282. Springer. m/s. 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466.
See section: Coronavirus Life Cycle—Assembly and Release
Unknown parameter|name-list-format=ignored (bantuan) - ^ Liu P, Shi L, Zhang W, He J, Liu C, Zhao C, dll. (November 2017). "Prevalence and genetic diversity analysis of human coronaviruses among cross-border children". Virology Journal (dalam bahasa Inggeris). 14 (1): 230. doi:10.1186/s12985-017-0896-0. PMC 5700739. PMID 29166910.
- ^ a b Forgie S, Marrie TJ (February 2009). "Healthcare-associated atypical pneumonia". Seminars in Respiratory and Critical Care Medicine. 30 (1): 67–85. doi:10.1055/s-0028-1119811. PMID 19199189.
- ^ phylogenetic tree of Beta-CoV[pautan mati kekal]
- ^ Corman VM, Muth D, Niemeyer D, Drosten C (2018). "Hosts and Sources of Endemic Human Coronaviruses". Advances in Virus Research. 100: 163–88. doi:10.1016/bs.aivir.2018.01.001. ISBN 978-0-12-815201-0. PMID 29551135.
Bacaan lanjut
sunting- Acheson, N. H. (2011). "Chapter 14: Coronaviruses". Fundamentals of molecular virology. Hoboken, NJ: John Wiley & Sons. m/s. 159–171. ISBN 9780470900598.
- Alwan A, Mahjour J, Memish ZA (2013). "Novel coronavirus infection: time to stay ahead of the curve". Eastern Mediterranean Health Journal. 19 Suppl 1: S3–4. PMID 23888787.
- Laude H, Rasschaert D, Delmas B, Godet M, Gelfi J, Charley B (June 1990). "Molecular biology of transmissible gastroenteritis virus". Veterinary Microbiology. 23 (1–4): 147–54. doi:10.1016/0378-1135(90)90144-K. PMID 2169670.
- Sola I, Alonso S, Zúñiga S, Balasch M, Plana-Durán J, Enjuanes L (April 2003). "Engineering the transmissible gastroenteritis virus genome as an expression vector inducing lactogenic immunity". Journal of Virology. 77 (7): 4357–69. doi:10.1128/JVI.77.7.4357-4369.2003. PMC 150661. PMID 12634392.
- Tajima M (1970). "Morphology of transmissible gastroenteritis virus of pigs. A possible member of coronaviruses. Brief report". Archiv für die Gesamte Virusforschung. 29 (1): 105–8. doi:10.1007/BF01253886. PMID 4195092.
Pautan luar
sunting| Wikimedia Commons mempunyai media berkaitan Koronavirus |
- Virus Pathogen Database and Analysis Resource (ViPR): Coronaviridae Diarkibkan 2013-03-12 di Wayback Machine
- German Research Foundation (Coronavirus Consortium)