Isositrat dehidrogenase
Isositrat dehidrogenase (IDH) (EC 1.1.1.42) dan (EC 1.1.1.41) ialah enzim yang memangkinkan dekarboksilasi oksidatif isositrat, menghasilkan alfa-ketoglutarat (α-ketoglutarat) dan CO2. Ini adalah proses dua langkah, yang melibatkan pengoksidaan isositrat (alkohol sekunder) kepada oksalosuksinat (keton), diikuti dengan penyahkarboksilan kumpulan karboksil beta kepada keton, membentuk alfa-ketoglutarat. Pada manusia, IDH wujud dalam tiga isobentuk: IDH3 memangkinkan langkah ketiga kitaran asid sitrik sambil menukar NAD+ kepada NADH dalam mitokondria. Isoform IDH1 dan IDH2 memangkinkan tindak balas yang sama di luar konteks kitaran asid sitrik, dan menggunakan NADP+ sebagai kofaktor berbanding NAD+. Ia terletak di sitosol serta mitokondria dan peroksisom.[2]
| Isositrat dehidrogenase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 | |||||||||
| Pengenal pasti | |||||||||
| Nombor EC | 1.1.1.42 | ||||||||
| Nombor CAS | 9028-48-2 | ||||||||
| Pangkalan data | |||||||||
| IntEnz | Lihat IntEnz | ||||||||
| BRENDA | Entri BRENDA | ||||||||
| ExPASy | Lihat NiceZyme | ||||||||
| KEGG | Entri KEGG | ||||||||
| MetaCyc | Laluan metabolik | ||||||||
| PRIAM | Profil | ||||||||
| Struktur PDB | RCSB PDB PDBj PDBe PDBsum | ||||||||
| Ontologi gen | AmiGO / EGO | ||||||||
| |||||||||
| Isositrat dehidrogenase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Pengenal pasti | |||||||||
| Nombor EC | 1.1.1.41 | ||||||||
| Nombor CAS | 9001-58-5 | ||||||||
| Pangkalan data | |||||||||
| IntEnz | Lihat IntEnz | ||||||||
| BRENDA | Entri BRENDA | ||||||||
| ExPASy | Lihat NiceZyme | ||||||||
| KEGG | Entri KEGG | ||||||||
| MetaCyc | Laluan metabolik | ||||||||
| PRIAM | Profil | ||||||||
| Struktur PDB | RCSB PDB PDBj PDBe PDBsum | ||||||||
| Ontologi gen | AmiGO / EGO | ||||||||
| |||||||||
| Isositrat dehidrogenase monomer | |||||||||
|---|---|---|---|---|---|---|---|---|---|
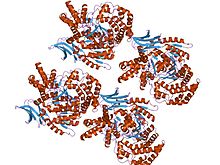 Monomer isositrat dehidrogenase monomer dengan kompleks dengan isositrat dan Mn2+ | |||||||||
| Pengenal pasti | |||||||||
| Simbol | IDH | ||||||||
| Pfam | PF03971 | ||||||||
| Klan Pfam | CL0270 | ||||||||
| InterPro | IPR004436 | ||||||||
| SCOP | 1ofg | ||||||||
| SUPERFAMILY | 1ofg | ||||||||
| |||||||||
Struktur sunting
NAD-IDH terdiri daripada 3 subunit yang dikawal secara alosterik, dan memerlukan ion Mg2+ atau Mn2+ bersepadu. Homolog terdekat yang mempunyai struktur yang diketahui ialah IDH bergantung kepada E. coli NADP yang hanya mempunyai 2 subunit dan 13% identiti dan 29% persamaan berdasarkan jujukan asid amino, menjadikannya berbeza dengan IDH manusia, dan tidak sesuai buat perbandingan rapat. Semua NADP-IDH yang diketahui ialah homodimer.
Kebanyakan isositrat dehidrogenase ialah dimer, dan secara khususnya homodimer (dua subunit monomer yang sama membentuk satu unit dimer). Dalam membandingkan C. glutamicum dan E. coli,[4] monomer dan dimer, masing-masing, kedua-dua enzim didapati "memangkinkan tindak balas yang serupa dengan cekap." Walau bagaimanapun, C. glutamicum direkodkan mempunyai 10 kali lebih banyak aktiviti daripada E. coli, dan tujuh kali lebih kekhususan terhadap NADP. C. glutamicum mengutamakan NADP+ berbanding NAD+. Dari segi kestabilan dengan tindak balas terhadap suhu, kedua-dua enzim mempunyai Tm atau suhu lebur yang sama pada kira-kira 55 °C hingga 60 °C. Walau bagaimanapun, monomer C. glutamicum menunjukkan kestabilan yang lebih konsisten dalam suhu yang lebih tinggi, satu hal yang dijangkakan. Dimer E. coli menunjukkan kestabilan pada suhu yang lebih tinggi daripada biasa disebabkan oleh interaksi antara dua subunit monomer.
Struktur ICDH-1 Mycobacterium tuberculosis (Mtb) yang terikat dengan NADPH dan Mn(2+) telah diselesaikan dengan kristalografi sinar-X. Ia merupakan homodimer, di mana setiap subunit mempunyai lipatan Rossmann, dan domain atas biasa bagi helaian β yang saling mengunci. ICDH-1 Mtb secara strukturnya hampir sama dengan ICDH manusia mutan R132H yang ditemui dalam astrositoma gred 4 CNS WHO, yang dahulunya dikelaskan[5] sebagai glioblastoma. Sama seperti R132H ICDH manusia, ICDH-1 Mtb juga memangkinkan pembentukan α-hidroksiglutarat.[6]
Kawal atur sunting
Langkah IDH kitaran asid sitrik selalunya (tetapi tidak selalu) tindak balas yang tidak dapat dipulihkan oleh kerana perubahan negatif besar dalam tenaga bebas. Oleh itu, ia mesti dikawal dengan teliti untuk mengelakkan kekurangan isositrat (dan oleh itu, pengumpulan alfa-ketoglutarat). Tindak balas dirangsang oleh mekanisme ketersediaan substrat ringkas (isositrat, NAD+ atau NADP+, Mg2+ / Mn2+), perencatan produk oleh NADH (atau NADPH di luar kitaran asid sitrik) dan alfa-ketoglutarat, dan maklum balas perencatan berpersaingan oleh ATP.[7] ncRNA yang dipelihara di hulu gen icd yang mengodkan IDH bergantungan NADP+ telah dilaporkan dalam genom bakteria kerana ciri-cirinya ncRNA ini menyerupai motif kawal atur sebelumnya yang dipanggil ribosuis, dan motif ncRNA icd-II telah dicadangkan sebagai calon ribosuis kuat.[8]
Mekanisme pemangkinan sunting
Isositrat dehidrogenase memangkinkan tindak balas kimia berikut:
- Isositrat + NAD+ 2-oksoglutarat + CO2 + NADH + H+
- Isositrat + NADP+ 2-oksoglutarat + CO2 + NADPH + H+[9][10][11]
Tenaga bebas keseluruhan untuk tindak balas ini ialah -8.4 kJ/mol.[12]
Langkah-langkah sunting
Dalam kitaran asid sitrik, isositrat yang dihasilkan daripada pengisomeran sitrat mengalami pengoksidaan dan penyahkarboksilan. Enzim isositrat dehidrogenase (IDH) memegang isositrat dalam tapak aktifnya dengan asid amino di sekelilingnya, termasuk arginina, tirosina, asparagina, serina, treonina dan asid aspartik.
Dalam rajah yang disediakan, kotak pertama menunjukkan tindak balas dehidrogenase isositrat keseluruhan. Bahan tindak balas yang diperlukan untuk mekanisme enzim ini ialah isositrat, NAD+ / NADP+, dan Mn2+ atau Mg2+. Hasil tindak balas ialah alfa-ketoglutarat, karbon dioksida, dan NADH + H+ / NADPH + H+.[10] Molekul air membantu menyahprotonkan atom oksigen isositrat.
Kotak kedua dalam rajah menggambarkan langkah 1 tindak balas, iaitu pengoksidaan alfa-karbon (C2 di sini, juga dipanggil alfa-C).[9][10] Dalam proses ini,[9] kumpulan alkohol alfa-karbon dinyahproton, dan pasangan elektron tunggal yang terhasil membentuk kumpulan keton pada karbon tersebut. NAD+/NADP+ bertindak sebagai kofaktor penerima elektron dan mengumpul hidrida yang terhasil daripada C2. Pengoksidaan karbon alfa memperkenalkan susunan molekul di mana elektron (dalam langkah seterusnya) akan mengalir dari kumpulan karboksil berdekatan, dan menolak elektron oksigen terikat berganda naik ke atas atom oksigen itu sendiri yang menarik proton daripada lisina berdekatan.
Kotak ketiga menggambarkan langkah 2, iaitu penyahkarboksilan oksalosuksinat. Dalam langkah ini,[9][10] oksigen kumpulan karboksil dinyahproton oleh tirosina berdekatan, dan elektron tersebut mengalir ke C2. Karbon dioksida, kumpulan keluar, terlepas daripada karbon beta isositrat (C3), dan elektron mengalir ke oksigen keton yang melekat di karbon alfa, memberikan cas negatif kepada atom oksigen yang berkaitan lalu membentuk ikatan berganda tak tepu alfa-beta antara karbon 2 dan 3.
Kotak keempat dan terakhir menggambarkan langkah 3, iaitu penepuan ikatan berganda tak tepu alfa-beta yang terbentuk dalam langkah sebelumnya. Oksigen bercas negatif (yang melekat pada alfa-karbon) menderma elektronnya, mengubah ikatan berganda keton dan menolak pasangan tunggal lain (yang membentuk ikatan ganda dua antara karbon alfa dan beta) "keluar" molekul. Pasangan tunggal ini pula mengambil proton daripada tirosina berhampiran.[14] Tindak balas ini menghasilkan pembentukan alfa-ketoglutarat, NADH + H+/NADPH + H+, dan CO2.
Mekanisme terperinci sunting
Dua sisa asid amino aspartat (di bawah kiri) berinteraksi dengan dua molekul air bersebelahan (w6 dan w8) dalam kompleks IDH babi isositrat Mn2+ untuk menyahproton alkohol daripada atom alfa-karbon. Pengoksidaan alfa-C juga berlaku dalam gambar ini, di mana NAD+ menerima hidrida yang menghasilkan oksalosuksinat. Bersama dengan perubahan stereokimia sp3 kepada sp2 di sekeliling alfa-C, terdapat kumpulan keton yang terbentuk daripada kumpulan alkohol. Pembentukan ikatan berganda keton ini membolehkan resonans berlaku apabila elektron yang turun dari kumpulan karboksilat yang meninggalkan bergerak ke arah keton.
Penyahkarboksilan oksalosuksinat (di bawah tengah) ialah langkah utama dalam pembentukan alfa-ketoglutarat. Dalam tindak balas ini, pasangan tunggal di hidroksil tiroksina bersebelahan mengabstrak proton daripada kumpulan karboksil.[14] Kumpulan karboksil ini juga dirujuk sebagai subunit beta dalam molekul isositrat. Penyahprotonan kumpulan karboksil menyebabkan pasangan elektron tunggal bergerak ke bawah menjadikan karbon dioksida, dan berpisah daripada oksalosuksinat. Elektron terus bergerak ke arah karbon alfa lalu menolak elektron ikatan berganda (membuat keton) sehingga mengabstrak proton daripada sisa lisina bersebelahan. Ikatan berganda tak tepu alfa-beta terhasil antara karbon 2 dan 3. Seperti dalam gambar, ion hijau mewakili sama ada Mg2+ atau Mn2+, kofaktor yang diperlukan dalam tindak balas ini. Ion logam membentuk satu kompleks kecil melalui interaksi ionik dengan atom oksigen pada karbon keempat dan kelima (juga dikenali sebagai subunit gama isositrat).
Selepas karbon dioksida berpecah daripada oksalosuksinat dalam langkah penyahkarboksilan (di bawah kanan), enol akan mentautomer menjadi bentuk keto. Pembentukan ikatan berganda keton dimulakan dengan penyahprotonan oksigen daripada karbon alfa (C#2) oleh lisina yang sama yang memprotonkan oksigen pada awalnya.[14] Pasangan elektron tunggal bergerak ke bawah, menendang pasangan tunggal yang membuat ikatan ganda dua. Pasangan elektron tunggal ini mengabstrakkan proton daripada tirosina yang menyahprotonkan kumpulan karboksil dalam langkah penyahkarboksilan. Sebab kita boleh berkata bahawa sisa Lys dan Tyr adalah yang sama daripada langkah sebelumnya adalah kerana ia membantu dalam menahan molekul isositrat dalam tapak aktif enzim. Kedua-dua residu ini akan dapat membentuk ikatan hidrogen bolak-balik selagi ia cukup dekat dengan substrat.[4]
Enzim dehidrogenase isositrat seperti yang dinyatakan di atas menghasilkan alfa-ketoglutarat, karbon dioksida, dan NADH + H+ /NADPH + H+. Terdapat tiga perubahan yang berlaku sepanjang tindak balas. Pengoksidaan karbon 2, penyahkarboksilan (kehilangan karbon dioksida) daripada karbon 3, dan pembentukan kumpulan keton dengan perubahan stereokimia daripada sp3 kepada sp2.[14]
Tapak aktif sunting
Struktur enzim isositrat dehidrogenase (IDH) dalam Escherichia coli ialah struktur ortolog IDH pertama yang dijelaskan dan difahami.[14] Sejak itu, struktur Escherichia coli IDH telah digunakan oleh kebanyakan penyelidik untuk membuat perbandingan dengan enzim isositrat dehidrogenase yang lain. Terdapat banyak pengetahuan terperinci tentang enzim bakteria ini, dan didapati bahawa kebanyakan dehidrogenase isositrat adalah serupa dalam struktur, dan oleh itu, juga dalam fungsi. Persamaan struktur dan fungsi ini memberikan alasan untuk mempercayai bahawa struktur itu dipelihara serta asid amino.[11] Oleh itu, tapak aktif dalam kalangan kebanyakan enzim dehidrogenase isositrat prokariot harus dipelihara juga, yang diperhatikan sepanjang banyak kajian yang dilakukan ke atas enzim prokariot. Enzim dehidrogenase isositrat eukariotik sebaliknya masih belum ditemui sepenuhnya. Setiap dimer IDH mempunyai dua tapak aktif.[14] Setiap tapak aktif mengikat molekul NAD+/NADP+ dan ion logam dwivalen (Mg2+, Mn2+). Secara amnya, setiap tapak aktif mempunyai urutan asid amino terpelihara untuk setiap tapak pengikatan tertentu. Dalam Desulfotalea psychrophila (IDH Dp)[14] dan babi (IDH Pc),[3] terdapat tiga substrat yang terikat pada tapak aktif.
- Isositrat mengikat di dalam tapak aktif terhadap jujukan terpelihara kira-kira lapan asid amino melalui ikatan hidrogen. Asid ini termasuk (mungkin berbeza dalam sisa tetapi dengan sifat yang serupa) tirosina, serina, asparagina, arginina, arginina, arginina, tirosina dan lisina. Kedudukan mereka di tulang belakang berbeza-beza tetapi mereka semua berada dalam jarak dekat (iaitu Arg131 DpIDH dan Arg133 PcIDH, Tyr138 DpIDH dan Tyr140 PcIDH).[14]
- Ion logam (Mg2+, Mn2+) mengikat terhadap tiga asid amino terpelihara melalui ikatan hidrogen. Asid amino ini termasuk tiga sisa aspartat.[14]
- NAD+ dan NADP+ mengikat dalam tapak aktif dalam empat kawasan dengan sifat yang sama antara enzim IDH. Wilayah ini berbeza-beza tetapi adalah sekitar [250–260], [280–290], [300–330] dan [365–380]. Sekali lagi, kawasan berbeza-beza tetapi kedekatan kawasannya dipelihara.[14]
Kepentingan klinikal sunting
Mutasi khusus dalam gen isositrat dehidrogenase IDH1 telah ditemui dalam beberapa jenis tumor,[16] terutamanya tumor otak termasuk astrositoma dan oligodendroglioma.[5] Pesakit dengan tumornya mempunyai mutasi IDH1 mempunyai kelangsungan hidup yang lebih lama berbanding pesakit yang tumornya mempunyai IDH1 jenis liar.[17][18] Tambahan pula, mutasi IDH2 dan IDH1 didapati dalam sehingga 20% daripada leukemia myeloid akut (AML) normal secara sitogenetik.[19][20] Mutasi ini diketahui menghasilkan D-2-hidroksiglutarat berbabding alfa-ketoglutarat.[21] D-2-hidroksiglutarat terkumpul dalam kepekatan yang sangat tinggi, menghalang fungsi enzim yang bergantung kepada alfa-ketoglutarat.[22] Ini membawa kepada keadaan hipermetilasi DNA dan histon, mengakibatkan ekspresi gen berbeza yang boleh mengaktifkan onkogen, dan menyahaktifkan gen penindas tumor. Akhirnya, ini boleh membawa kepada jenis kanser yang diterangkan di atas.[23] Mutasi mozek somatik gen ini juga didapati dikaitkan dengan penyakit Ollier dan sindrom Maffucci.[24] Walau bagaimanapun, kajian baru-baru ini juga menunjukkan bahawa D-2-hidroksiglutarat boleh ditukar kembali kepada alfa-ketoglutarat sama ada dengan enzim atau tidak.[25][26] Kajian lanjut diperlukan untuk memahami sepenuhnya peranan mutasi IDH1 (dan D-2-hidroksiglutarat) dalam kanser. Penyelidikan terkini menyerlahkan mutasi penyebab kanser dalam isositrat dehidrogenase yang boleh menyebabkan pengumpulan metabolit D-2-hidroksiglutarat (D-2HG). Notarangelo et al. menunjukkan bahawa kepekatan D-2HG yang tinggi boleh bertindak sebagai perencat langsung laktat dehidrogenase dalam sel T tikus. Perencatan enzim metabolik ini mengubah metabolisme glukosa dalam sel T dan menghalang percambahannya, pengeluaran sitokin, dan keupayaan untuk membunuh sel sasaran.[27]
Isozim sunting
Berikut ialah senarai isozim isositrat dehidrogenase manusia:
Bergantungan NADP+ sunting
Setiap isozim bergantungan NADP+ berfungsi sebagai homodimer:
|
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Bergantungan NAD+ sunting
Isozim isositrat dehidrogenase 3 ialah heterotetramer yang terdiri daripada dua subunit alfa, satu subunit beta, dan satu subunit gama:
|
|
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Rujukan sunting
- ^ PDB: 1CW7; Cherbavaz DB, Lee ME, Stroud RM, Koshland DE (January 2000). "Active site water molecules revealed in the 2.1 A resolution structure of a site-directed mutant of isocitrate dehydrogenase". Journal of Molecular Biology. 295 (3): 377–385. doi:10.1006/jmbi.1999.3195. PMID 10623532.
- ^ "Peroxisomal NADP-Dependent Isocitrate Dehydrogenase. Characterization and Activity Regulation during Natural Senescence". Plant Physiology. 121 (3): 921–928. November 1999. doi:10.1104/pp.121.3.921. PMC 59455. PMID 10557241.
- ^ a b c d e f PDB: 1LWD; "Crystal structure of porcine mitochondrial NADP+-dependent isocitrate dehydrogenase complexed with Mn2+ and isocitrate. Insights into the enzyme mechanism". The Journal of Biological Chemistry. 277 (45): 43454–43462. November 2002. doi:10.1074/jbc.M207306200. PMID 12207025.
- ^ a b "A highly specific monomeric isocitrate dehydrogenase from Corynebacterium glutamicum". Archives of Biochemistry and Biophysics. 383 (2): 238–245. November 2000. doi:10.1006/abbi.2000.2082. PMID 11185559.
- ^ a b "The 2021 WHO Classification of Tumors of the Central Nervous System: a summary". Neuro-Oncology. 23 (8): 1231–1251. August 2021. doi:10.1093/neuonc/noab106. PMC 8328013 Check
|pmc=value (bantuan). PMID 34185076. Unknown parameter|displayauthors=ignored (bantuan) - ^ "Structural, kinetic and chemical mechanism of isocitrate dehydrogenase-1 from Mycobacterium tuberculosis". Biochemistry. 52 (10): 1765–1775. March 2013. doi:10.1021/bi400037w. PMC 3706558. PMID 23409873.
- ^ "Role of a peroxisomal NADP-specific isocitrate dehydrogenase in the metabolism of the riboflavin overproducer Ashbya gossypii". Journal of Molecular Catalysis B: Enzymatic. 10 (1–3): 335–343. 2000. doi:10.1016/S1381-1177(00)00135-1.
- ^ "Comprehensive discovery of novel structured noncoding RNAs in 26 bacterial genomes". RNA Biology. 18 (12): 2417–2432. December 2021. doi:10.1080/15476286.2021.1917891. PMC 8632094 Check
|pmc=value (bantuan). PMID 33970790. Unknown parameter|displayauthors=ignored (bantuan) - ^ a b c d Begley TP, McMurry J (2005). The Organic Chemistry of Biological Pathways. Roberts and Co. Publishers. m/s. 189–190. ISBN 0-9747077-1-6.
- ^ a b c d Cox M, Nelson DR, Lehninger AL (2005). Lehninger Principles of Biochemistry. San Francisco: W.H. Freeman. m/s. 609–611. ISBN 0-7167-4339-6.
- ^ a b "Crystal structure of the monomeric isocitrate dehydrogenase in the presence of NADP+: insight into the cofactor recognition, catalysis, and evolution". The Journal of Biological Chemistry. 278 (38): 36897–36904. September 2003. doi:10.1074/jbc.M304091200. PMID 12855708.
- ^ Garrett R, Grisham CM (2012). Biochemistry. Cengage Learning. m/s. 621. ISBN 978-1133106296.
- ^ "Nondecarboxylating and decarboxylating isocitrate dehydrogenases: oxalosuccinate reductase as an ancestral form of isocitrate dehydrogenase". Journal of Bacteriology. 190 (6): 2050–2055. March 2008. doi:10.1128/JB.01799-07. PMC 2258884. PMID 18203822.
- ^ a b c d e f g h i j k l m "Structural and functional properties of isocitrate dehydrogenase from the psychrophilic bacterium Desulfotalea psychrophila reveal a cold-active enzyme with an unusual high thermal stability". Journal of Molecular Biology. 372 (1): 130–149. September 2007. doi:10.1016/j.jmb.2007.06.040. PMID 17632124.
- ^ "Monoclonal antibody specific for IDH1 R132H mutation". Acta Neuropathologica. 118 (5): 599–601. November 2009. doi:10.1007/s00401-009-0595-z. PMID 19798509.
- ^ "Clinical development of IDH1 inhibitors for cancer therapy". Cancer Treatment Reviews. 103: 102334. February 2022. doi:10.1016/j.ctrv.2021.102334. PMID 34974243 Check
|pmid=value (bantuan). Unknown parameter|displayauthors=ignored (bantuan) - ^ "Patients with IDH1 wild type anaplastic astrocytomas exhibit worse prognosis than IDH1-mutated glioblastomas, and IDH1 mutation status accounts for the unfavorable prognostic effect of higher age: implications for classification of gliomas" (PDF). Acta Neuropathologica. 120 (6): 707–718. December 2010. doi:10.1007/s00401-010-0781-z. PMID 21088844. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "The combination of IDH1 mutations and MGMT methylation status predicts survival in glioblastoma better than either IDH1 or MGMT alone". Neuro-Oncology. 16 (9): 1263–1273. September 2014. doi:10.1093/neuonc/nou005. PMC 4136888. PMID 24510240. Unknown parameter
|displayauthors=ignored (bantuan) - ^
"The common feature of leukemia-associated IDH1 and IDH2 mutations is a neomorphic enzyme activity converting alpha-ketoglutarate to 2-hydroxyglutarate". Cancer Cell. 17 (3): 225–234. March 2010. doi:10.1016/j.ccr.2010.01.020. PMC 2849316. PMID 20171147. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "WT1 recruits TET2 to regulate its target gene expression and suppress leukemia cell proliferation". Molecular Cell. 57 (4): 662–673. February 2015. doi:10.1016/j.molcel.2014.12.023. PMC 4336627. PMID 25601757. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "Cancer-associated IDH1 mutations produce 2-hydroxyglutarate". Nature. 465 (7300): 966. June 2010. Bibcode:2010Natur.465..966D. doi:10.1038/nature09132. PMC 3766976. PMID 20559394. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "The oncometabolite 2-hydroxyglutarate inhibits histone lysine demethylases". EMBO Reports. 12 (5): 463–469. May 2011. doi:10.1038/embor.2011.43. PMC 3090014. PMID 21460794. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "The driver and passenger effects of isocitrate dehydrogenase 1 and 2 mutations in oncogenesis and survival prolongation". Biochimica et Biophysica Acta (BBA) - Reviews on Cancer. 1846 (2): 326–341. December 2014. doi:10.1016/j.bbcan.2014.05.004. PMID 24880135.
- ^ "Ollier disease and Maffucci syndrome are caused by somatic mosaic mutations of IDH1 and IDH2". Nature Genetics. 43 (12): 1262–1265. November 2011. doi:10.1038/ng.994. PMID 22057236. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "Non-enzymatic chemistry enables 2-hydroxyglutarate-mediated activation of 2-oxoglutarate oxygenases". Nature Communications. 5: 3423. March 2014. Bibcode:2014NatCo...5.3423T. doi:10.1038/ncomms4423. PMC 3959194. PMID 24594748. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "Transformation by the (R)-enantiomer of 2-hydroxyglutarate linked to EGLN activation". Nature. 483 (7390): 484–488. February 2012. Bibcode:2012Natur.483..484K. doi:10.1038/nature10898. PMC 3656605. PMID 22343896. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "Oncometabolite d-2HG alters T cell metabolism to impair CD8+ T cell function". Science. 377 (6614): 1519–1529. September 2022. doi:10.1126/science.abj5104. PMC 9629749 Check
|pmc=value (bantuan). PMID 36173860 Check|pmid=value (bantuan). Unknown parameter|displayauthors=ignored (bantuan)